«`html
DynamicBind: глубокий подход к динамическому докингу белка-лиганда и поиску лекарств
Современные методы в области предсказания структур белков сталкиваются с проблемами в представлении их динамики, учитывающей влияние лигандов. Это критично для понимания функций белков и развития лекарственных средств. Традиционные методы докинга часто ограничиваются рассмотрением белков как жестких структур, что снижает их точность. Молекулярно-динамические симуляции могут предложить соответствующие конформации белков, но они требуют значительных вычислительных ресурсов. Недавние достижения, такие как AlphaFold, предсказывают структуры по последовательностям, но генерируют только несколько конформаций, упуская динамическую природу белков, что сказывается на точности докинга. Исследователи из Galixir Technologies, School of Pharmaceutical Science, Sun Yat-sen University, Center for Theoretical Biological Physics and Department of Chemistry, Rice University, и Global Institute of Future Technology, Shanghai Jiao Tong University разработали метод DynamicBind, использующий глубокое обучение для предсказания конформаций, специфичных для лигандов, из структур несвязанных белков без обширного сэмплирования. DynamicBind отличается высокой точностью в докинге и виртуальном скрининге, способен учесть значительные изменения конформаций белков и отыскать скрытые углубления в новых целевых белках. Этот метод показывает потенциал в ускорении разработки малых молекул для ранее недоступных целей и продвижении вычислительного поиска лекарственных средств.
Основные особенности DynamicBind
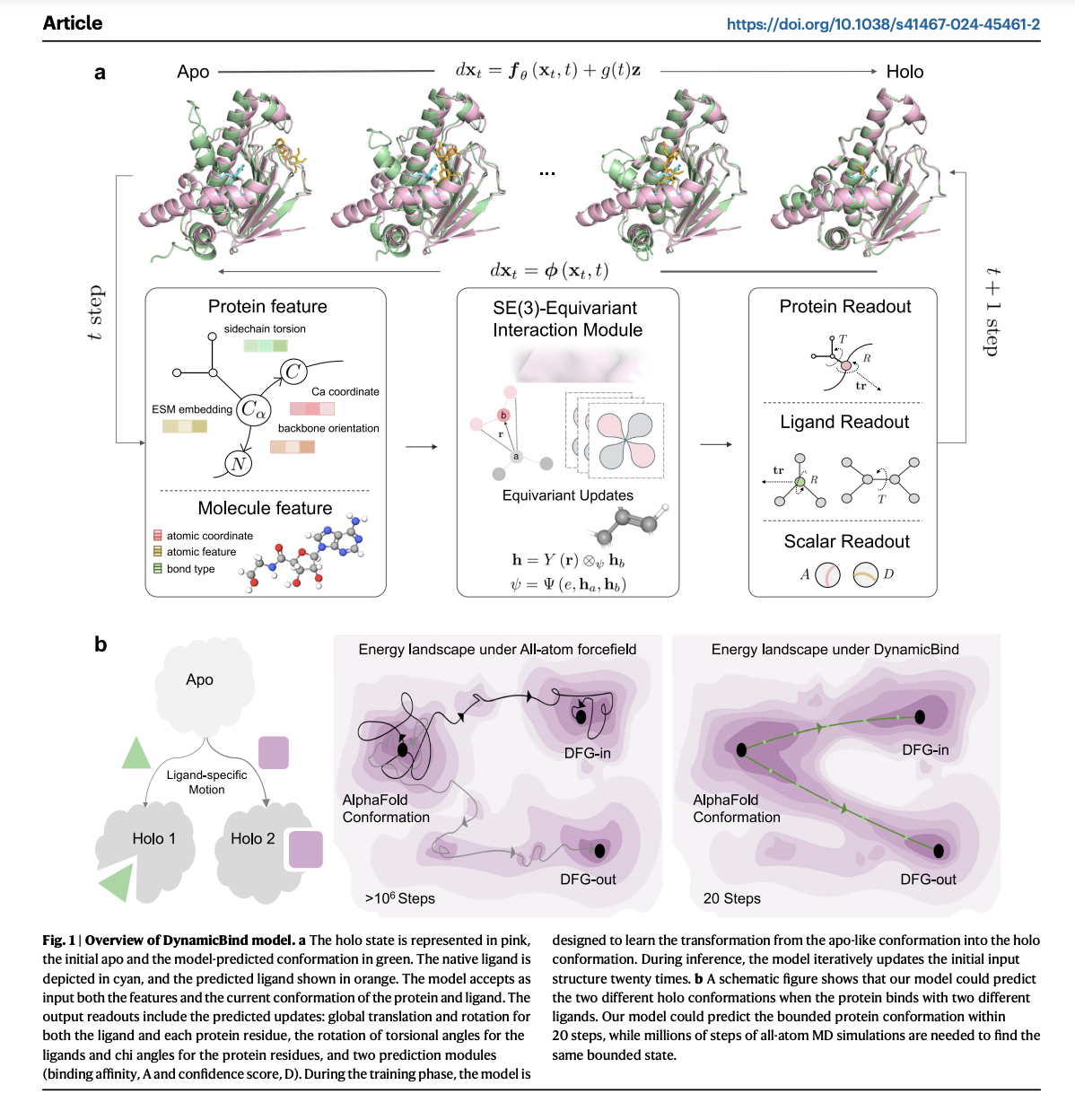
DynamicBind – это универсальный инструмент для предсказания структур белково-лигандных комплексов, способный адаптироваться к значительным изменениям конформаций белков. В процессе вывода модель постепенно корректирует положения лигандов и внутренние углы на протяжении 20 итераций, а также приспосабливает конформации белков, в частности углы боковых цепей, улучшая предсказания AlphaFold. В отличие от традиционных моделей, он использует морфоподобное преобразование вместо гауссовских шумовых воздействий, что улучшает способность модели улавливать биологически значимые изменения конформаций. DynamicBind превосходит в предсказании положения лигандов, уменьшая столкновения и выявляя скрытые углубления, демонстрируя свой потенциал для приложений в области поиска лекарственных средств.
В заключение, DynamicBind интегрирует генерацию конформаций белков и предсказание положения лигандов в единую глубокую систему обучения, значительно превосходя традиционные молекулярно-динамические симуляции по скорости. В отличие от обычных методов докинга, требующих заранее определенных связывающих углублений, DynamicBind выполняет глобальный докинг, что идеально подходит для обнаружения скрытых углублений, снижая возможные побочные эффекты за счет направленного воздействия на конкретные белки. Хотя модель проявляет отличные характеристики, требуются улучшения для более общего использования на белках с низкой гомологией последовательностей. Новые достижения в области крио-электронной микроскопии и включение данных о связывающей способности могут усилить потенциал DynamicBind.
«`