«`html
Улучшение прогнозирования пространственной экспрессии генов с помощью мета-алгоритмической интеграции SPRITE
Пространственно-распределенная одноклеточная транскриптомика предоставляет информацию о экспрессии генов в тканях, но текущие технологии ограничены способностью измерять только небольшое количество генов. Для решения этой проблемы были разработаны алгоритмы для предсказания или дополнения экспрессии дополнительных генов. Эти методы часто используют сопоставленные данные одноклеточного секвенирования РНК, объединяя пространственные и РНК-последовательности для предсказаний. Однако большинству подходов все еще нужно полностью использовать информацию о взаимосвязи между генами (например, коэкспрессия) или клетками (например, пространственная близость). Внедрение этой взаимосвязанной информации может улучшить точность прогнозирования экспрессии генов и улучшить последующие биологические анализы.
Практические решения и ценность
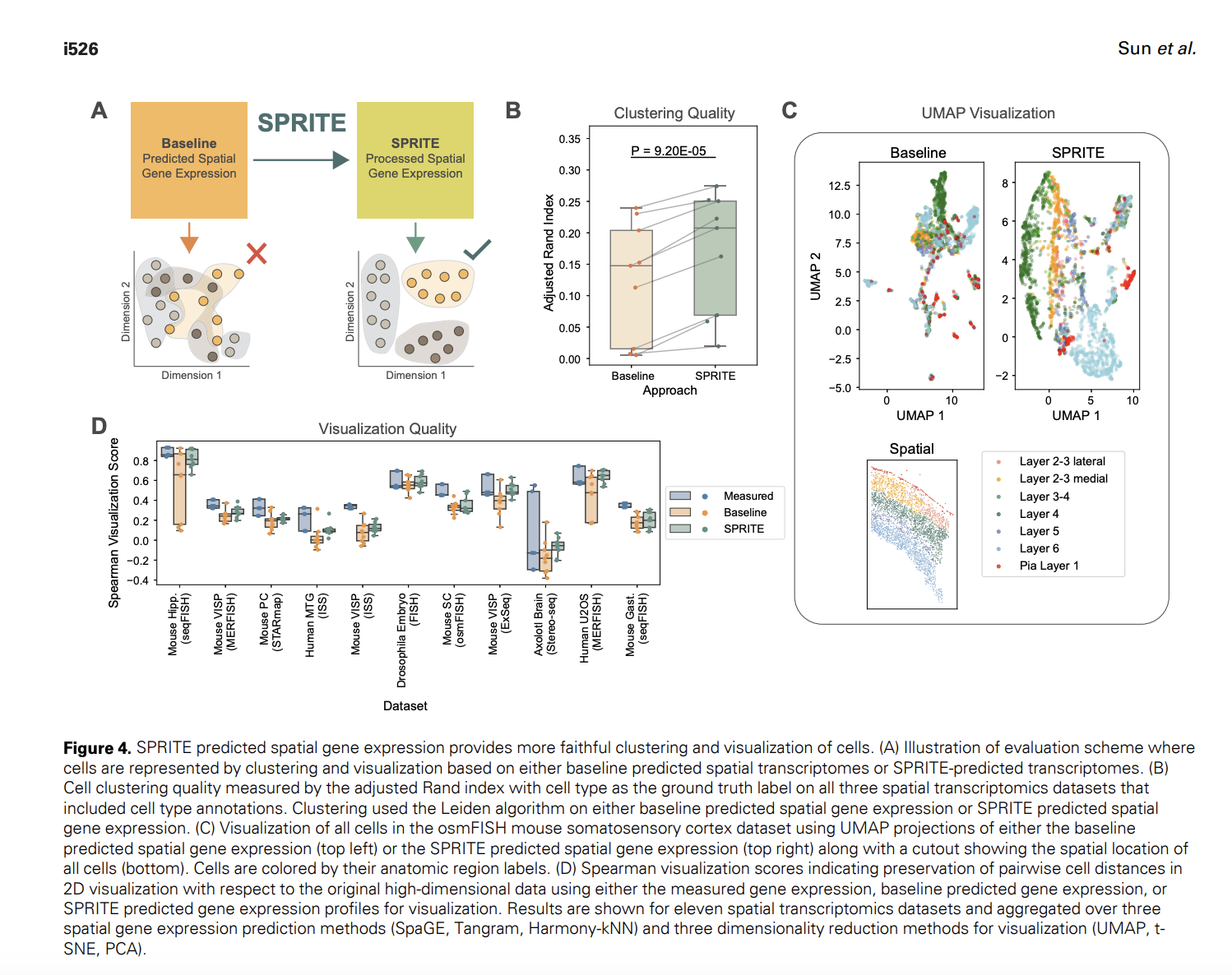
Исследователи из Университета Стэнфорда и Гарвардского университета разработали мета-алгоритм SPRITE (Spatial Propagation and Reinforcement of Imputed Transcript Expression), предназначенный для улучшения прогнозирования пространственной экспрессии генов. SPRITE улучшает прогнозы существующих методов, распространяя информацию по сетям корреляции генов и пространственных графиков соседства. Этот двухэтапный процесс повышает точность прогнозов пространственной экспрессии генов, что приводит к лучшей производительности в последующих анализах, таких как кластеризация клеток, визуализация и классификация. SPRITE можно интегрировать в анализ данных пространственной транскриптомики для улучшения качества выводов на основе прогнозируемой экспрессии генов.
Алгоритм SPRITE был протестирован на одиннадцати эталонных наборах данных, объединяющих пространственную транскриптомику с данными РНК-секвенирования из четырех видов: человека, мыши, мухи-доядов, и аксолотля. Эти наборы данных, использующие различные технологии и типы тканей, были выбраны для поддержания последовательности внутри видов и категорий тканей. В рамках использования данные РНК-секвенирования прошли нормализацию и логарифмическое преобразование. SPRITE был оценен с использованием трех методов прогнозирования пространственной экспрессии генов — SpaGE, Tangram и Harmony-kNN, каждый из которых использует различные подходы для сопоставления и прогнозирования экспрессии генов. Точность этих прогнозов до и после применения SPRITE измерялась с использованием PCC и средней абсолютной ошибки (MAE).
«`
Please let me know if you need further assistance.